POLYPHASIC IDENTIFICATION AND WHOLE-GENOME ANALYSIS OF A SELF-FLOCCULATING BACTERIAL STRAIN, MASSILIA sp. W12
-
摘要:
研究以筛选自絮凝菌作为生物絮团系统外源强化菌株为目的, 从中华草龟养殖池塘中分离纯化出一株自絮凝菌株, 对其进行多相分类学鉴定确定W12菌株的分类地位, 基因注释分析探讨其作为强化菌株的可行性。W12菌株与Massilia arenosa MC02T相似性最高, 为96.21%, W12菌株与其亲缘关系较近的几株模式菌株的ANI值在71.61%—72.23%, dDDH值在19.6%—22.9%, 远低于判别细菌新种的阈值, 进一步表明W12为一株马赛菌属新种。此外, 全基因组分析发现W12的自絮凝机制可能与喜树脂动胶菌相似, 由胞外多糖和PEP-CTERM蛋白质共同介导, 推测菌株W12通过PHB合成途径Ⅰ涉及PhaA、PhaB和PhaC三种酶来合成PHB。研究从中华草龟养殖池塘分离到的自絮凝菌株W12是一株马赛菌属新种, W12菌株可以合成PHB, 由胞外多糖和PEP-CTERM蛋白质共同介导其自絮凝, 此性能与生物絮团的形成息息相关。通过基因组分析对菌株的潜在应用价值进行挖掘, 以期为生物絮团系统的功能强化提供候选菌株。
-
关键词:
- 多相分类 /
- 自絮凝 /
- 胞外多糖 /
- PEP-CTERM蛋白质 /
- 马赛菌
Abstract:The functional components of bioflocs are highly complex microbial aggregates, and the design and optimization of these intricate microbial communities are crucial for improving biofloc performance. To screen self-flocculating bacteria as exogenous reinforcement strains for biofloc systems, a self-flocculating strain was isolated and purified from the breeding pond of Chinese grass turtles. Its taxonomic status was identified through polyphasic taxonomy, which included 16S rRNA gene sequencing, phylogeneticanalysis, and whole genome sequence analyses. Strain W12 showed the highest similarity with Massilia arenosa MC02T at 96.21%, the ANI values for strain W12 ranged from 71.61% to 72.23%, and the dDDH values ranged from 19.6% to 22.9% with several type strains of closely related species, which were much lower than the thresholds for discriminating a new bacteria species. This strongly indicates that W12 is a novel species within the genus Massilia. In addition, genome-wide analysis reveales that the self-flocculation mechanism of W12 may is similar to that of the Zoogloea resiniphila, likely mediated by a combination of extracellular polysaccharides and PEP-CTERM proteins. It is hypothesized that strain W12 synthesizes PHB through the PHB synthesis pathway Ⅰ, involving three enzymes: PhaA, PhaB, and PhaC. The discovery of strain W12, a novel species of the genus Massilia isolated from a turtle breeding pond, highlights its ability to synthesize PHB and self-flocculation through extracellular polysaccharides and PEP-CTERM proteins. These properties are significant for bioflocs formation, underscoring the potential application of strains in enhancing the biofloc system.
-
全国渔业经济统计公报的最新数据显示, 2022年, 全国水产品总产量6865.91万吨, 养殖产量高达5565.46万吨, 是捕捞产品产量的4倍[1]。高密度的养殖伴随而来的水资源的过度使用、养殖排污、饲料浪费及成本增加等问题[2, 3], 使得当下水产养殖业面临质量提升和绿色发展的双重挑战。生物絮团技术(Biofloc technology, BFT)作为一种新型的水产养殖生产系统, 被认为是解决水产养殖业现有问题的一种新技术[4]。典型的生物絮团是由菌胶团或丝状细菌为骨架, 附着细菌分泌的胞外聚合物或由静电引力作用将微藻、残饵、粪便等颗粒有机物絮凝在一起自然形成的絮团状聚合物[5]。生物絮团的功能主体是高度复杂的微生物聚集体, 通过对目标菌群的定向调控或添加外源菌株来构建功能性生物絮团, 可有效提高生物絮团养殖系统的生物强化效能和系统稳定性。
微生物细胞有两种差异显著的形态: 即常规的单细胞游离态和多细胞自絮凝形成的聚集体。聚集体通常有两种不同的表现方式: 在液体–空气或是液体–固体表面呈生物膜(Biofilm)形态的聚集和在液体中自发沉降呈絮凝(Floc)状态的聚集[6]。细菌自絮凝表现为细胞自发聚集成团并沉降到溶液底部, 与游离细菌相比, 自絮凝细菌在实际应用中具有更明显的优势。有研究表明自絮凝菌株在生物絮团系统中也有发现, 且提高碳氮比可显著提高自絮凝菌株的占比[7]。自絮凝菌株的显著特征是可以产生由多糖等生物大分子组成的胶状胞外聚合物(EPS), 而EPS在生物絮团的形成中起着至关重要的作用, 对生物颗粒的黏附过程和微生物生理过程有重要影响[8]。
自絮凝菌株作为外源强化菌株因其良好的沉降性能和吸附性能, 比其他益生菌更容易在生物絮团系统内定植。本研究以筛选自絮凝菌作为生物絮团系统外源强化菌株为目的, 从中华草龟养殖池塘中分离纯化出一株自絮凝菌株, 对其进行多相分类学鉴定及基因组信息分析, 挖掘絮凝相关的功能基因, 以期为生物絮团系统的功能强化提供候选菌株。
1. 材料与方法
1.1 材料
菌株 样品采自安徽省芜湖市某水产养殖场的中华草龟养殖池塘水体, 在R2A培养基上利用稀释涂布平板法进行菌株的分离纯化, 筛选获得的W12菌株现保藏于中国典型培养物保藏中心, 保藏编号为CCTCC AB 2021126。
培养基 R2A培养基: 酵母提取物0.5 g, 蛋白胨0.5 g, 酪蛋白水解物0.5 g, 葡萄糖 0.5 g, 可溶性淀粉0.5 g, K2HPO4 0.3 g, MgSO4 0.024 g, 丙酮酸钠 0.3 g, 琼脂 20 g, 加水至1000 mL, pH 7.0—7.4; 121℃灭菌20min。
TSA培养基: 蛋白胨15 g, 大豆胨5 g, NaCl 5 g, 琼脂15-20 g, 加水至1000 mL, pH 7.2。
LB培养基: 酵母提取物5 g, 蛋白胨10 g, NaCl 5 g, 加水至1000 mL, pH 7.0—7.2。
主要试剂和仪器 2.5%戊二醛固定液购自上海麦克林生化科技股份有限公司, 其他常规化学试剂均为国产分析纯购自国药集团化学试剂有限公司, 分子生物学所用的酶、引物等试剂购自生工生物工程(上海)股份有限公司。恒温振荡培养箱(ZQTY-90S, 上海知楚仪器有限公司)、PCR仪及凝胶成像仪(Bio-Rad公司), 透射电子显微镜(日立公司)。
1.2 菌落和菌体形态观察鉴定
将菌株W12划线接种于R2A琼脂培养基, 28℃培养2d进行菌落形态观察。从划线培养基中挑取菌落, 接种于R2A液体培养基中, 28℃、180 r/min培养2d, 以备革兰氏染色及透射电镜观察菌体形态, 具体方法参照文献[9]。
1.3 生理生化特征测定
为测定菌株最适温度、最适pH、盐度耐受特征, 将1% W12菌株种子液接种至R2A液体培养基中, 在相应温度(4、10、15、20、28、30、35、40、45和50℃)下, 180 r/min培养7d; 相应pH范围(pH 5—10, 间隔1个单位)和NaCl浓度(0.05%、0.1%、0.15%、0.2%、0.5%、1和2%; w/v)条件下, 28℃、180 r/min培养7d, 观察菌株生长情况。使用API 20NE试剂盒检测部分生化实验和糖同化实验。
1.4 化学分类分析
活化好的菌株接种到R2A液体培养基中, 28℃、180 r/min培养2d, 离心收集菌体, 然后用无菌水洗涤2—3次, 冷冻干燥后获得的菌体送至广州微生物研究所进行呼吸醌、脂肪酸、极性脂的鉴定。用高效液相色谱来分析醌型。采用Agilent 6890色谱仪参照WJA07-2002技术标准进行细菌脂肪酸鉴定。采用双相点样展层法分析极性脂。
1.5 分子生物学分析
细菌基因组的提取参照DNA提取试剂盒说明书进行。用提取的菌株基因组DNA作为模板, 以细菌通用引物27F (5′AGAGTTTGATCCTGGCTCAG-3′)和1492R (5′-TACGGCTACCTTGTTACGACTT-3′)进行16S rRNA基因序列的扩增。PCR反应体系: 模板DNA 1 μL, 上下游引物各1 μL, Premix Taq 20 μL, ddH2O 17 μL。PCR反应程序: 95℃ 5min; 95℃ 30s, 55℃ 30s, 72℃ 40s, 30次循环; 72℃ 5min。将扩增后的DNA产物克隆到pMD19-T 载体上, 转化到E.coli (DH5α)感受态细胞中, 挑取阳性克隆送生工生物工程(上海)股份有限公司进行一代测序。所测序列经拼接后与EzBioCloud数据库中的模式菌株进行16S rRNA基因序列相似性分析, 使用MEGA X软件用邻接法构建系统发育树, bootstrap自展1000次检验系统发育树拓扑结构置信区间。
菌株的全基因组测序委托上海美吉生物技术有限公司采用Illumina+PacBio的测序方式完成, 基因组完成图利用组装软件unicycler v0.4.8进行三代序列组装。使用NCBI原核基因组自动注释程序(https://www.ncbi.nlm.nih.gov/genomes/static/Pipeline.html)和RAST网站(https://rast.nmpdr.org/rast.cgi)对基因组进行注释[10]。根据预测蛋白序列与KEEG数据库、NR数据库、COG数据库比对后的结果, 分析菌株自絮凝及产PHB相关的功能基因。使用EzGenome Web服务器(http://www.ezbiocloud.net/tools/ani)的OrthoANI算法计算菌株W12与相近菌株的平均核苷酸一致性(ANI)值, 来分析其基因组相关性[11]。通过Genome-to-Genome Distance Calculator(GGDC)网站(http://ggdc.dsmz.de/distcalc2.php)进行数字DNA-DNA杂交值(dDDH)的计算[12]。利用Circos软件绘制基因组圈图。
2. 结果
2.1 菌株的鉴定
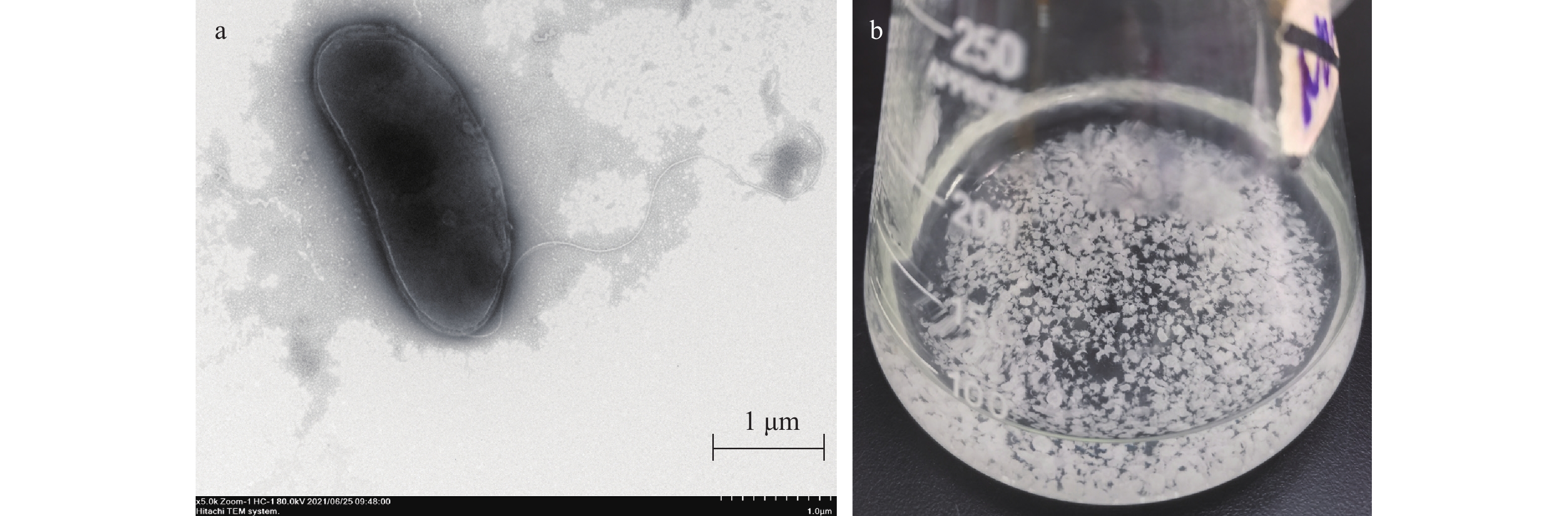
形态学观察 W12菌株属革兰氏阴性细菌, 在R2A琼脂平板上呈乳白色、圆形、边缘规则, 不透明。细胞呈杆状、单极鞭毛、不产芽孢(图 1a), 菌株在R2A液体培养基中自发形成聚集体沉降(图 1b)。
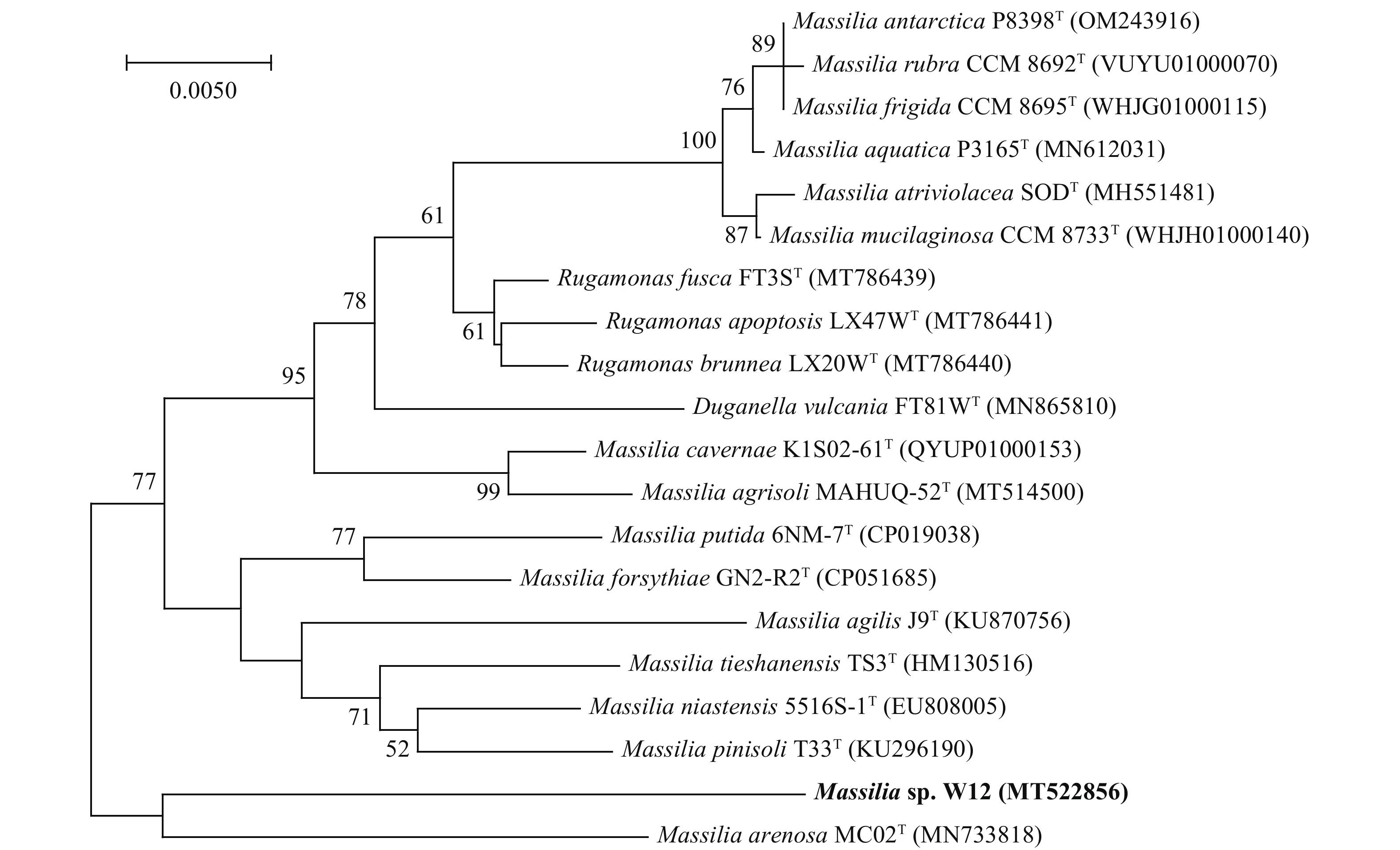
分子生物学鉴定 W12菌株的16S rRNA基因序列长度为1482 bp, 已上传GenBank, 登录号为MT522856。利用EzBioCloud服务器进行16S rRNA基因序列相似性分析, 结果发现与W12菌株相似性最高的模式菌株为Massilia arenosa MC02T, 相似性为96.21%, 研究者们常以菌种的16S rRNA基因序列与模式菌株相似性低于98.7%作为划分新种的阈值[13], 菌株W12可能是潜在的新种。采用邻接法构建系统发育进化树, 如图 2所示, W12菌株与Massilia属的其他菌株聚在一起, 同时与Massilia arenosa MC02T菌株的遗传距离最近, 说明菌株W12属于马赛菌属, 菌株W12在系统进化树中独立成一个分支, 表明W12菌株可能是Massilia 属内的一个新种。
为进一步确定W12菌株的分类地位, 基于全基因组测序数据, 进行了dDDH和ANI计算分析。如表 1所示, W12菌株与其亲缘关系较近的几株模式菌株dDDH值在19.6%—22.9%, ANI值在71.61%—72.23%, 远低于dDDH与ANI判别细菌新种的阈值70%[14]和 95%[15]。一般认为DNA-DNA杂交值在20%—70%的为同一属, 进一步说明菌株W12为Massilia属内的新种。
表 1 菌株W12与最相似模式菌株分子生物学特征比较Table 1. Comparison of the molecular characteristics in the strain W12 and the most similar type strains最相似菌株
Most similar strain基因相似性
16S rRNA gene
similarity (%)平均核苷酸
一致性值
ANI value (%)杂交值
dDDH value
DNA-DNA (%)Massilia arenosa MC02T 96.21 72.14 19.6 Rugamonas apoptosis LX47WT 96.21 72.02 19.9 Massilia albidiflava 45T 95.7 72.03 21.4 Massilia putida 6NM-7T 96 71.61 22.9 Massilia atriviolacea SODT 95.86 72.23 19.9 2.2 菌株的生理生化特征
W12菌株在LB和TSA培养基上不能生长。菌株能够在15—45℃, pH 5—9和0—0.05%氯化钠浓度下生长良好, 其最适生长条件分别为28℃, pH 7.0和0%氯化钠浓度。在API 20NE系统中, β-葡萄糖苷酶和蛋白酶阳性, 其他反应均呈阴性。
2.3 菌株的化学分类特征
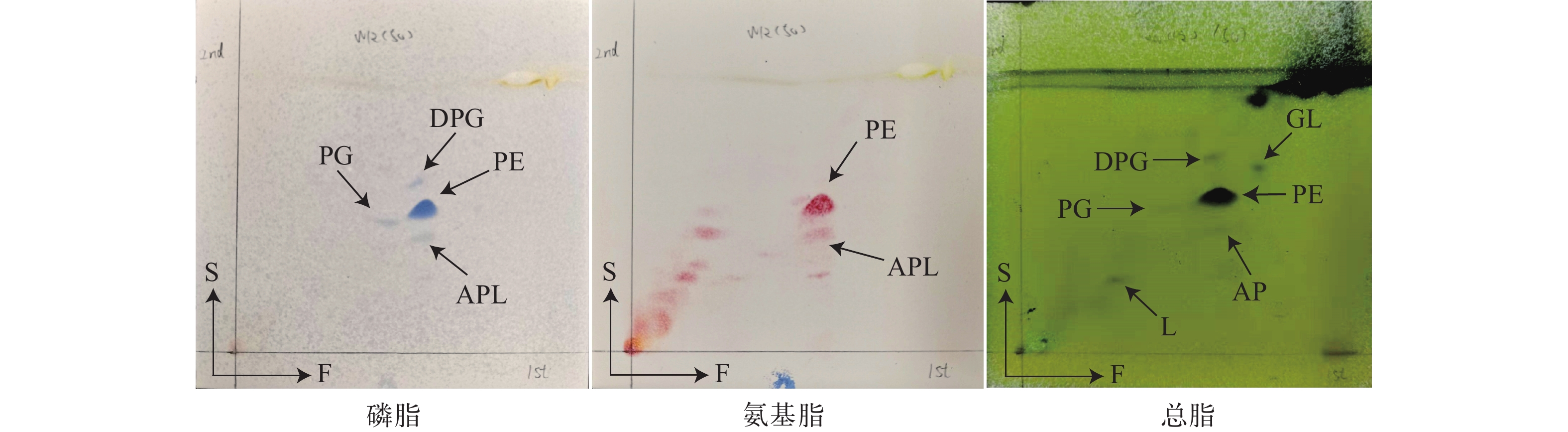
表 2所示, 菌株W12的主要脂肪酸(>5%)组成为C16:0 (30.14%), 综合特征3 (C16:1ω7c和/或C16:1ω6c)(57.21%), 与其相似菌株在脂肪酸组成和含量上存在些差异, 但主要脂肪酸成分相同, 且符合马赛菌属特征, 但是与Rugamonas属典型菌株的脂肪酸可以明显区分开。双相薄层层析显示(图 3), 菌株W12的极性脂主要为二磷脂酰甘油(Diphosphatidylglycerol, DPG)、磷脂酰乙醇胺(Phosphatidylethanolamine, PE)、磷脂酰甘油(Phosphatidylglycerol, PG)、未知的磷脂(Unidentified lipid)、未知的糖脂(Unidentified glycolipid)和未知的氨基脂(Unidentified aminophospholipid)。菌株W12的特征磷脂是DPG、PG和PE, 符合马赛菌属特征。菌株W12以Q-8为主要的呼吸醌型。综上, 菌株W12符合马赛菌属的特征, 而又与其相似菌具有明显区别。
表 2 W12菌株与相似菌株脂肪酸组成比较Table 2. Comparison of fatty acid composition in strain W12 and similar strainsFatty acid W12 Massilia
arenosa
MC02TRugamonas
rivuli
FT103WTMassilia
agilis J9TC12﹕0 2.93 Tr 11 5.5 C14﹕0 Tr 5.64 Tr 3.3 C16﹕0 30.14 30.01 26.5 36.7 C16﹕1ω7c ND ND 52.7 ND C18﹕1ω7c ND ND 4.5 4.6 Iso-C16﹕0 Tr Tr ND 1.4 C10﹕0 3-OH 4.35 5.26 4.4 4.3 C14﹕0 2-OH ND ND ND 2.1 Summed Feature 3 57.21 49.07 ND 34.7 Summed Feature 8 3.34 5.65 ND ND Note: *Summed features represent groups of two or three fatty acids that cannot be separated by the Microbial Identification System; Summed feature 3 consist of C16﹕1ω7c and/or C16﹕1ω6c; summed feature 8 consist of C18﹕1 ω7c and/or C18﹕1 ω6c; ND. not detected; Tr Fatty acids comprising <1% of the total; Data for strain W12 are obtained from this study, data for Massilia arenosa MC02T[16], Rugamonas rivuli FT103WT[17], and Massilia agilis J9T[18] are obtained from the cited references 2.4 菌株W12的基因组特征
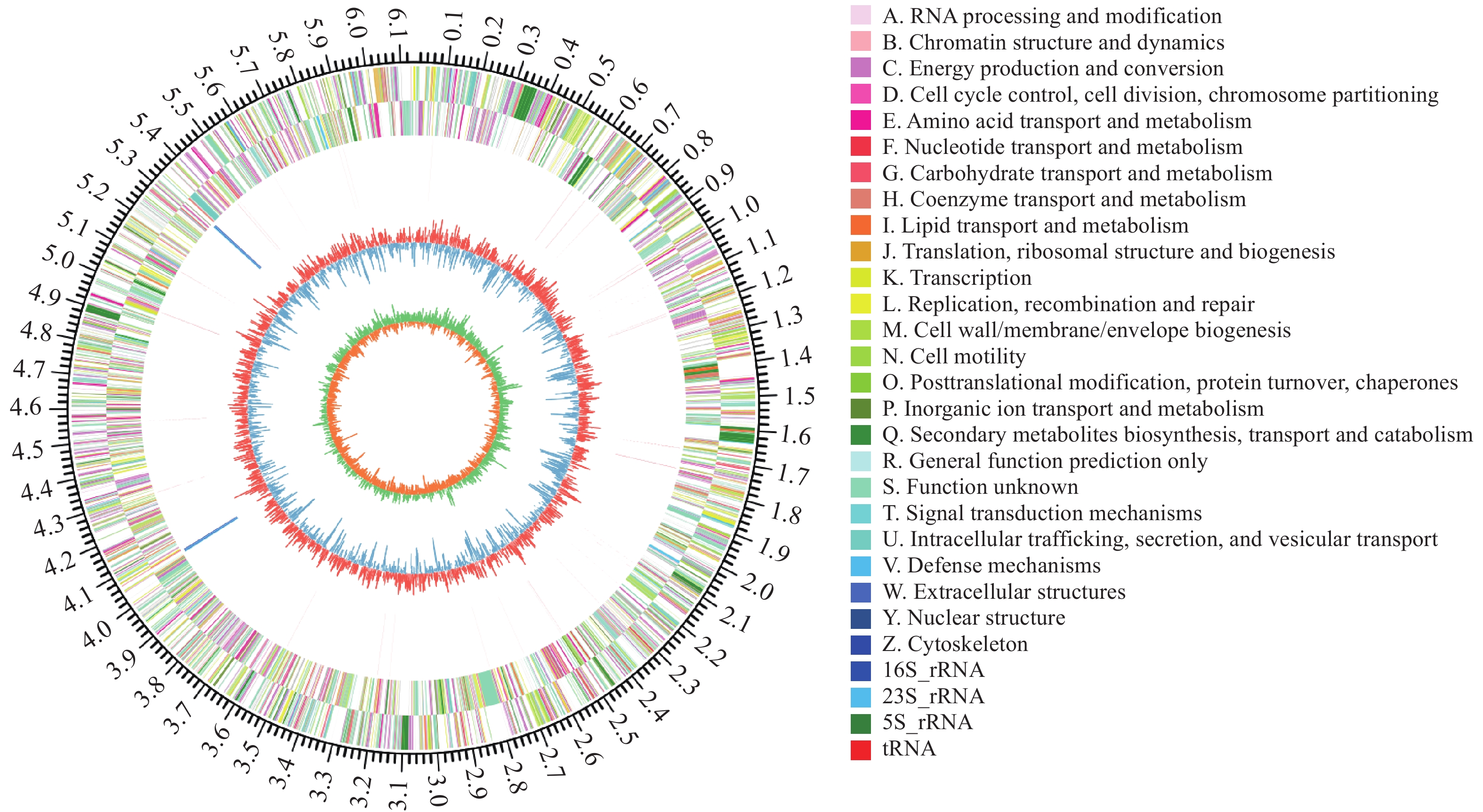
菌株W12的基因组序列已在美国生物信息中心(NCBI)GenBank数据库提交, 登录号为CP147776。菌株W12的全基因组大小为6136891 bp, G+C含量57.24 mol%, 低于马赛菌属G+C含量(62.4—69.4) mol%[19], 也低于目前已报道的Rugamonas属菌株的G+C含量[17], W12基因组中未检测到质粒。蛋白编码基因为5584个, tRNA编码基因69个, rRNAs编码基因12个(4个5S rRNA、4个16S rRNA、4个23S rRNA), 基因组中有20个基因岛, 总长度为355884 bp, 平均长度为17794 bp。预测有1个前噬菌体, 总长度为19283 bp。在基因组中鉴定到27个CRISPRs。基因组圈图显示, W12菌株的染色体为环状基因组, 圈图的最外面一圈表明基因组的大小为6136891 bp; 第2圈和第3圈为正链、负链上的编码序列, 不同颜色的编码序列表示COG的功能分类不同, 编码基因总数量5584个, 其中可以注释到COG的基因数3564个, 包括氨基酸的运输和代谢、无机盐离子的运输和代谢等; 第4圈为rRNA和tRNA; 第5圈为GC含量; 最内一圈为GC-Skew值(图 4)。
COG数据库注释结果显示, 3564个基因完成蛋白注释, 占全部基因的72.82%。其中信号转导机制相关基因得到最高注释量, 达到402个, 有184个未知功能基因, 主要功能预测和氨基酸转运及代谢基因数量分别为334和296个, 细胞壁/膜/包膜生物发生的基因数量为275个, 参与翻译、核糖体结构和生物发生的基因为269个。
碳水化合物相关酶数据库(CAZy)中共注释到111个基因, 糖基转移酶(Glycosyl Transferases)注释到的基因数量最多, 为40个, 显著地高于其他分类。其次是糖苷水解酶(Glycoside Hydrolases)和糖类酯解酶(Carbohydrate Esterases), 分别涉及26个和25个基因。
2.5 菌株W12自絮凝相关基因注释
本研究前期利用分子遗传学和基因组学手段, 对喜树脂动胶菌(Zoogloea resiniphila MMB)和申氏杆菌(Shinella zoogloeoides ATCC19623T)自絮凝的分子机制加以研究, 发现一个约40 kb的胞外多糖生物合成大型基因簇及PEP-CTERM蛋白参与喜树脂动胶菌自絮凝[20, 21], 纤维素合成基因簇参与申氏杆菌自絮凝[22]。PEP-CTERM蛋白的典型特征是其氨基端具有典型的分泌信号肽, 羧基端具有保守的PEP-CTERM域, 包括PEP (Pro-Glu-Pro, 脯氨酸-谷氨酸-脯氨酸)基序及带电的残基尾巴[23]。
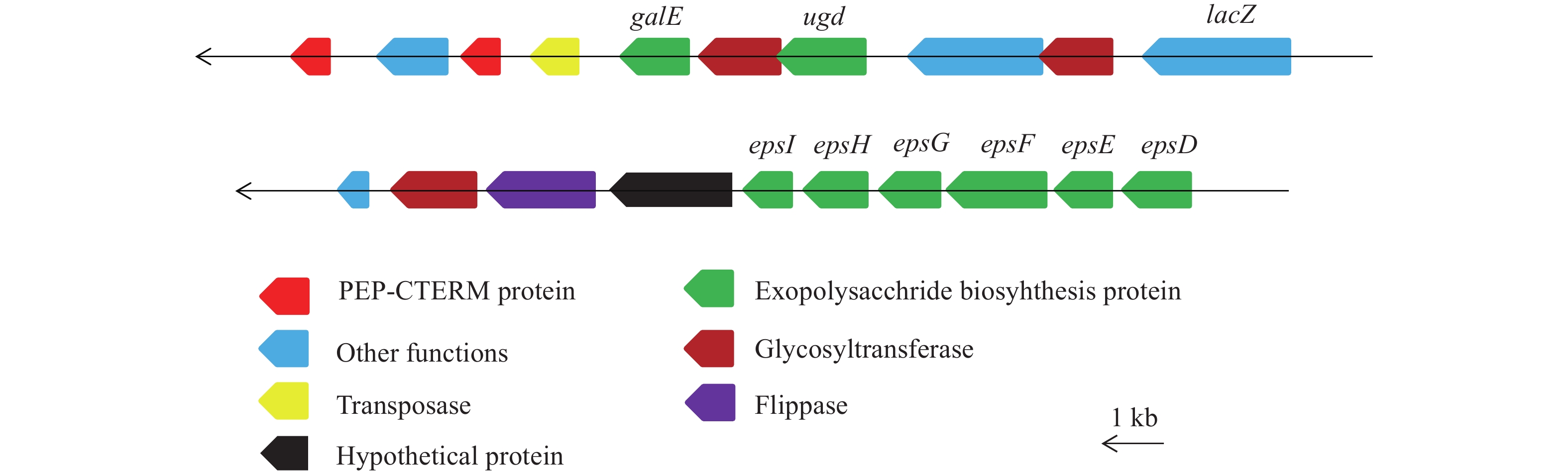
通过基因组注释和BLAST分析发现, W12基因组中拥有30个编码PEP-CTERM结构域蛋白质的基因, 其中2个pep-cterm基因与胞外多糖合成相关的基因簇连锁。如图 5所示, 该基因簇中有3个编码糖基转移酶的基因, 编码UDP-葡萄糖4-差向异构酶和UDP-葡萄糖6-脱氢酶的基因及胞外多糖链长、重复单元合成、聚合和输出等基因。前期研究发现PEP-CTERM蛋白富含天冬酰胺(缩写为Asn或者N)残基, 可能与胞外多糖通过N-连锁的糖基化形成复合物, 包裹微生物细胞群体来介导菌株自絮凝[21]。Haft等[23]从结构类比推测胞外多糖生物合成基因簇所编码的EpsH蛋白可能是革兰氏阴性细菌中的外分选酶(Exosortase), 作为一个8次穿膜的膜蛋白, EpsH可能像革兰氏阳性菌的分选酶一样具有转肽酶功能, 对PEP-CTERM结构域进行切割并且短暂结合到自身的活性位点半胱氨酸上, 然后再进行接下来的进一步分选和加工步骤。菌株W12基因组中未发现纤维素合成相关的基因, 因此W12自絮凝的分子机制可能与喜树脂动胶菌相似。
2.6 菌株W12 PHB合成途径预测
聚羟基丁酸酯(Polyhydroxybutyrate, PHB)是一种具有代表性的短链聚羟基脂肪酸酯(Polyhydroxyalkanoates, PHA), 脂溶性颗粒, 具有疏水性, 且在高温下稳定, 对水生动物生长性能、消化功能、抗氧化能力、免疫功能及肠道菌群等方面均有一定的影响, 适用于饲料加工和水产养殖环境中[24]。研究表明生物絮团的形成与PHB在细菌内大量聚集所需条件一致, 且PHB是生物絮团中的重要活性成分, 含量约为5%—20%[25]。表 3中列出了通过各个数据库相互比对筛选出的菌株W12 基因组中涉及PHB代谢相关的编码基因, 在菌株W12中, PHB生物合成涉及3种酶: PhaA、PhaB和PhaC, PHB生物合成始于通过不同途径产生的两个乙酰辅酶A分子, 被β-酮硫解酶(PhaA)缩合成乙酰乙酰-CoA, 乙酰乙酰-CoA还原酶(PhaB)将乙酰乙酰-CoA还原成3-羟基丁酰-CoA, 最后, 3-羟基丁酰-CoA单体通过PHA合成酶 (PhaC)聚合成PHB, 菌株W12的 PHB合成途径与任佳楠等[26]报道的Massilia sp. UMI-21相似。
表 3 W12基因组中与PHB合成代谢相关的基因Table 3. Genes associated with PHB anabolism in the genome W12基因
Gene蛋白
ProteinDNA链
DNA chain基因位置
Gene location基因大小
Gene length (bp)phaA/phbA/atoB β-酮硫解酶 β-ketothiolase — 1536402—
15376041203 — 4817680—
48189091230 — 5852691—
58538691179 phaB/phbB 乙酰乙酰辅酶A还原酶Acetoacetyl-CoA reductase — 5851841—
5852581741 phaC/phbC PHA聚合酶 PHA synthase + 2020701—
2020985285 + 2158638—
21604881851 — 5334054—
53357571704 phaR PHA合成调控蛋白
PHA synthesis regulator protein+ 2160647—
2161201555 phaZ PHA解聚酶 PHA depolymerase + 3803239—
38044741236 3. 讨论
本研究从中华草龟养殖池塘分离出一株马赛菌属W12, 并使用多相分类学方法确定了其分类地位。菌株W12的主要脂肪酸为综合特征3 (C16:1ω7c和/或C16﹕1ω6c)和C16﹕0, 极性脂主要有DPG、PG和PE, 以Q-8为主要的呼吸醌型。化学成分分析显示菌株W12符合马赛菌属的特征, 且16S rRNA基因序列相似性比对显示菌株W12与Massilia arenosa MC02T相似度较高, 相似性为96.21%, 在系统发育树中, 菌株W12与马赛菌属的菌株聚在一起, 说明W12属于马赛菌属, 但是其独立成为一支, 说明W12可能是马赛菌属的一个新种。基于全基因组序列分析发现, W12菌株与其亲缘关系较近的几株模式菌株ANI值在71.61%—72.23%, dDDH值在19.6%—22.9%, 均低于新种判别的界限值, 进一步确定W12菌株是马赛菌属内的新种。
基因组分析发现菌株W12的自絮凝机制与喜树脂动胶菌相似, 由胞外多糖和PEP-CTERM蛋白质共同介导。生物絮团技术来源于污水处理工艺中的活性污泥法, 接种自絮凝微生物已成为生物强化好氧颗粒污泥快速形成与稳定的策略之一[27], 而自絮凝细菌在生物絮团技术中的应用报道较少。菌株W12基因组中注释到PHB合成代谢相关的基因, 推测PHB的合成途径与Massilia sp. UMI-21相似, 涉及PhaA、PhaB和PhaC三种酶, 为典型的PHB合成途径1。综上, 菌株W12可以产生胞外多糖, 也可以合成PHB, 这些性能与生物絮团的形成息息相关。后期将菌株W12作为生物絮团系统的外源强化菌株, 对缩短絮团培养时间、发挥絮团水质净化功能及提高絮团营养价值可能具有重要的作用。自絮凝菌株因其自发沉降能力相较于游离菌株在工业发酵及环境修复领域具有明显的应用优势, 后续对菌株W12的特殊功能进行深入研究, 挖掘其潜在的应用价值, 对工业应用也具有重要意义。
-
表 1 菌株W12与最相似模式菌株分子生物学特征比较
Table 1 Comparison of the molecular characteristics in the strain W12 and the most similar type strains
最相似菌株
Most similar strain基因相似性
16S rRNA gene
similarity (%)平均核苷酸
一致性值
ANI value (%)杂交值
dDDH value
DNA-DNA (%)Massilia arenosa MC02T 96.21 72.14 19.6 Rugamonas apoptosis LX47WT 96.21 72.02 19.9 Massilia albidiflava 45T 95.7 72.03 21.4 Massilia putida 6NM-7T 96 71.61 22.9 Massilia atriviolacea SODT 95.86 72.23 19.9 表 2 W12菌株与相似菌株脂肪酸组成比较
Table 2 Comparison of fatty acid composition in strain W12 and similar strains
Fatty acid W12 Massilia
arenosa
MC02TRugamonas
rivuli
FT103WTMassilia
agilis J9TC12﹕0 2.93 Tr 11 5.5 C14﹕0 Tr 5.64 Tr 3.3 C16﹕0 30.14 30.01 26.5 36.7 C16﹕1ω7c ND ND 52.7 ND C18﹕1ω7c ND ND 4.5 4.6 Iso-C16﹕0 Tr Tr ND 1.4 C10﹕0 3-OH 4.35 5.26 4.4 4.3 C14﹕0 2-OH ND ND ND 2.1 Summed Feature 3 57.21 49.07 ND 34.7 Summed Feature 8 3.34 5.65 ND ND Note: *Summed features represent groups of two or three fatty acids that cannot be separated by the Microbial Identification System; Summed feature 3 consist of C16﹕1ω7c and/or C16﹕1ω6c; summed feature 8 consist of C18﹕1 ω7c and/or C18﹕1 ω6c; ND. not detected; Tr Fatty acids comprising <1% of the total; Data for strain W12 are obtained from this study, data for Massilia arenosa MC02T[16], Rugamonas rivuli FT103WT[17], and Massilia agilis J9T[18] are obtained from the cited references 表 3 W12基因组中与PHB合成代谢相关的基因
Table 3 Genes associated with PHB anabolism in the genome W12
基因
Gene蛋白
ProteinDNA链
DNA chain基因位置
Gene location基因大小
Gene length (bp)phaA/phbA/atoB β-酮硫解酶 β-ketothiolase — 1536402—
15376041203 — 4817680—
48189091230 — 5852691—
58538691179 phaB/phbB 乙酰乙酰辅酶A还原酶Acetoacetyl-CoA reductase — 5851841—
5852581741 phaC/phbC PHA聚合酶 PHA synthase + 2020701—
2020985285 + 2158638—
21604881851 — 5334054—
53357571704 phaR PHA合成调控蛋白
PHA synthesis regulator protein+ 2160647—
2161201555 phaZ PHA解聚酶 PHA depolymerase + 3803239—
38044741236 -
[1] 农业农村部渔业渔政管理局. 2022中国渔业统计年鉴 [M]. 北京: 中国农业出版社, 2022.] Fisheries and Fisheries Administration Bureau of the Ministry of Agriculture and Rural Affairs. China Fishery Statistical Yearbook 2022 [M]. Beijing: China Agriculture Press, 2022. [
[2] Su H C, Hu X J, Xu Y, et al. Persistence and spatial variation of antibiotic resistance genes and bacterial populations change in reared shrimp in South China [J]. Environment International, 2018(119): 327-333.
[3] Thitamadee S, Prachumwat A, Srisala J, et al. Review of current disease threats for cultivated penaeid shrimp in Asia [J]. Aquaculture, 2016(452): 69-87.
[4] Zhao Y P, Xue B R, Bi C W, et al. Influence mechanisms of macro-infrastructure on micro-environments in the recirculating aquaculture system and biofloc technology system [J]. Reviews in Aquaculture, 2022, 15(3); 991-1009.
[5] Avnimelech Y. Biofloc Technology-A Practical Guide Book (2nd ed) [M]. Baton Rouge: the World Aquaculture Society, 2012: 73-91.
[6] Davey M E, O'toole G A. Microbial biofilms: from ecology to molecular genetics [J]. Microbiology and Molecular Biology Reviews, 2000, 64(4): 847-867. doi: 10.1128/MMBR.64.4.847-867.2000
[7] Deng M, Li L, Dai Z L, et al. Aerobic denitrification affects gaseous nitrogen loss in biofloc-based recirculating aquaculture system [J]. Aquaculture, 2020(529): 735686.
[8] Abakari G, Wu X, He X, et al. Bacteria in biofloc technology aquaculture systems: roles and mediating factors [J]. Reviews in Aquaculture, 2022, 14(3): 1260-1284. doi: 10.1111/raq.12649
[9] 李萍, 黄文丽, 熊川等. 一株耐镉细菌Exiguobacterium acetylicum TC13的分离鉴定及全基因组分析 [J]. 微生物学通报, 2024, 51(5): 1583-1599.] Li P, Huang W L, Xiong C, et al. Isolation, identification, and whole-genome analysis of a cadmium-tolerant bacterium Exiguobacterium acetylicum TC13 [J]. Microbiology China, 2024, 51(5): 1583-1599. [
[10] Overbeek R, Olson R, Pusch G D, et al. The SEED and the rapid annotation of microbial genomes using Subsystems Technology (RAST) [J]. Nucleic Acids Research, 2014, 42(D1): D206-D214. doi: 10.1093/nar/gkt1226
[11] Yoon S H, Ha S M, Lim J, et al. A large-scale evaluation of algorithms to calculate average nucleotide identity [J]. Antonie van Leeuwenhoek, 2017, 110(10): 1281-1286. doi: 10.1007/s10482-017-0844-4
[12] Meier-Kolthoff J P, Auch A F, Klenk H P, et al. Genome sequence-based species delimitation with confidence intervals and improved distance functions [J]. BMC Bioinformatics, 2013(14): 60.
[13] Yarza P, Yilmaz P, Pruesse E, et al. Uniting the classification of cultured and uncultured bacteria and archaea using 16S rRNA gene sequences [J]. Nature Reviews Microbiology, 2014, 12(9): 635-645. doi: 10.1038/nrmicro3330
[14] Auch A F, Von Jan M, Klenk H P, et al. Digital DNA-DNA hybridization for microbial species delineation by means of genome-to-genome sequence comparison [J]. Standards in Genomic Sciences, 2010, 2(1): 117-134. doi: 10.4056/sigs.531120
[15] Jain C, Rodriguez-R L M, Phillippy A M, et al. High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries [J]. Nature Communications, 2018, 9(1): 5114. doi: 10.1038/s41467-018-07641-9
[16] Raths R, Peta V, Bucking H. Massilia arenosa sp. nov., isolated from the soil of a cultivated maize field [J]. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(6): 3912-3920.
[17] Lu H B, Deng T C, Liu F F, et al. Rugamonas aquatica sp. nov. and Rugamonas rivuli sp. nov., isolated from a subtropical stream in PR China [J]. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(5): 3328-3334.
[18] Altankhuu K, Kim J. Massilia solisilvae sp. nov., Massilia terrae sp. nov. and Massilia agilis sp. nov., isolated from forest soil in South Korea by using a newly developed culture method [J]. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(8): 3026-3032.
[19] 杨恩东, 崔丹曦, 汪维云. 马赛菌属细菌研究进展 [J]. 微生物学通报, 2019, 46(6): 1537-1548.] Yang E D, Cui D X, Wang W Y. Research progress on the genus Massilia [J]. Microbiology China, 2019, 46(6): 1537-1548. [
[20] An W X, Guo F, Song Y L, et al. Comparative genomics analyses on EPS biosynthesis genes required for floc formation of Zoogloea resiniphila and other activated sludge bacteria [J]. Water Research, 2016(102): 494-504.
[21] Gao N, Xia M, Dai J C, et al. Both widespread PEP-CTERM proteins and exopolysaccharides are required for floc formation of Zoogloea resiniphila and other activated sludge bacteria [J]. Environmental Microbiology, 2018, 20(5): 1677-1692. doi: 10.1111/1462-2920.14080
[22] Gao N, Dai J C, Liu Y Q, et al. Cellulose-mediated floc formation by the activated sludge bacterium Shinella zoogloeoides ATCC 19623 [J]. BMC Microbiology, 2022, 22(1): 104. doi: 10.1186/s12866-022-02516-y
[23] Haft D H, Paulsen I T, Ward N, et al. Exopolysaccharide-associated protein sorting in environmental organisms: the PEP-CTERM/EpsH system. Application of a novel phylogenetic profiling heuristic [J]. BMC Biology, 2006(4): 29.
[24] 刘玉, 隋丽英, 邓元告, 等. 聚β-羟基丁酸酯(PHB)对中华绒螯蟹(Eriocheir sinensis)幼蟹生长和肝胰腺酶活力的影响 [J]. 海洋与湖沼, 2013, 44(5): 1333-1338.] Liu Y, Sui L Y, Deng Y G, et al. Effect of poly-hydroxybutyrate on growth and hepatopancreatic enzyme activities of Eriocheir sinensis juveniles [J]. Oceanologia et Limnologia Sinica, 2013, 44(5): 1333-1338. [
[25] Ruan Y J, Zhu L, Xu X Y. Study on the flocs poly-β-hydroxybutyrate production and process optimization in the bio-flocs technology system [J]. Bioresource Technology, 2011, 102(16): 7599-7602. doi: 10.1016/j.biortech.2011.05.028
[26] 任佳楠. Massilia sp. UMI-21 PHA合成途径中相关酶鉴定及其对PHA合成的影响 [D]. 长春: 长春理工大学, 2021: 32-36.] Ren J N. Identification of related enzymes in PHA synthesis pathway and their influence on PHA synthesis of Massilia sp. UMI-21 [D]. Changchun: Changchun University of Science and Technology, 2021: 32-36. [
[27] Han X S, Jin Y, Yu J G. Rapid formation of aerobic granular sludge by bioaugmentation technology: A review [J]. Chemical Engineering Journal, 2022, 437(1): 1385-8947.



 下载:
下载: